浅谈统计方法的重要性
前言:前段时间,因疫苗问题闹得沸沸扬扬,这是非常严重的问题,关乎民生,国家应该彻查。因违法惩罚偏低,使得部分奸商为利铤而走险,不顾百姓健康。常言道,出来混的,迟早要还!我愿相信国家会在法制上想法制止这种损人利己的投机倒把。国人好斗,早已深入人心,诸如四大名著,哪部不言斗?啥时国人能跟数据好好斗斗?如果数据是原料,统计方法就是菜谱。理论上,给定一些原料,应该会得到一份合适的最佳菜谱。同理,对于给定一份数据,理论上也应有最合适的统计方法。不同统计方法,得到的结果可能会相差万里。因此,若要从事严谨的科研生产,应当关注相关行业的最新统计方法。懒散得随意分析生产数据,是一种罪过,也是对事业的不尊重,还是对人生的不尊重。今天演示的空间分析,即会看到最佳菜谱的重要性。
空间分析(spatial analysis)现已成为农林业遗传评估的常见方法之一,但其最早提出者竟是一位医生1,这出乎我的意料。话说1840s末期,伦敦爆发了严重的霍乱疫情,死了不少人。主流理论认为霍乱通过“肮脏的空气”传染的,这错误理论导致霍乱未能得到有效控制。伦敦皇家外科医学院有位医生,名叫John Snow,虽然年纪轻轻但已事业有成,他不认同霍乱传染的主流理论,他认为水里携带的细菌才是主因。他用几年时间,走遍伦敦进行详细调研和病情记录,绘制了如下的点图:
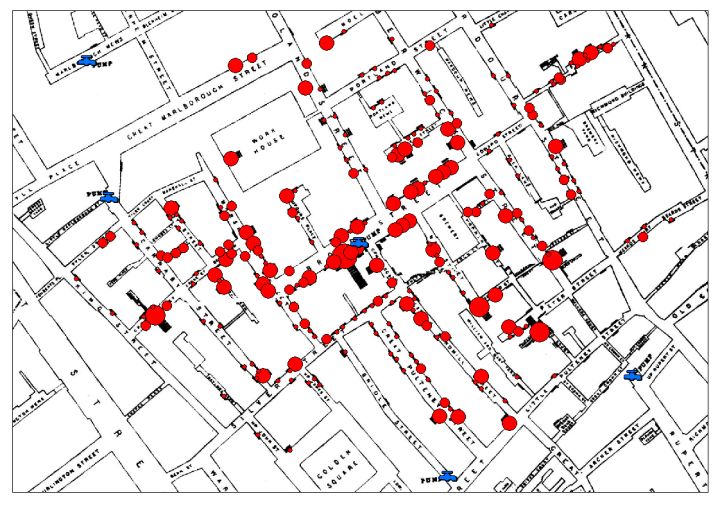
最后,Snow将传染源锁定在一个公共抽水机,并说服政府将抽水机挪走。因为当时泰晤士河已被污染,然后通过抽水机传播霍乱。当年Snow锁定的抽水机,现已成了伦敦的地标之一:

Snow调查表明疾病的发病率跟地理位置是相关的,这就是空间分析方法的始祖。可惜,Snow英年早逝,四十多岁就累死了。
牛人已去,历史继续。Snow肯定没想到他的方法,被后人广泛运用到诸多行业,比如农林业上。现在介绍农林业上应用的空间分析原理及其示例。
1 空间分析的基本原理
遗传评估分析的通用混合线性模型如下: $$y=xb+zu+e$$
对于空间分析2,将上式中的误差e进一步分解为e = ξ + η。ξ为空间相关的误差,η为空间不相关的随机误差。η反应了微环境差异、非加性遗传效应以及测量误差。
Authur等(1997)将ξ设为行、列的自回归AR1$\otimes$AR1,因此其方差协方差矩阵为:
$$Var(\xi) = \sigma_\xi^2 [Σ_c(ρ_c) \otimes Σ_r(ρ_r)]$$
其中,$\otimes$为矩阵的Kronecker积(kronecker product),$Σ_c(ρ_c)、Σ_r (ρ_r)$为列、行的回归矩阵,列、行自回归参数分别为$ρ_c、ρ_r$。
$Σ_c(ρ_c)$回归矩阵具体如下:
$$ \left( \begin{array}{ccc} 1 & ρ_c & ρ_c^2 & \ldots & ρ_c^{c-1} \\ ρ_c & 1 & ρ_c & \ldots & \vdots \\ ρ_c^2 & ρ_c & 1 & \ldots & \vdots \\ \vdots & \vdots & \vdots & \ddots & \vdots \\ ρ_c^{c-1} & \vdots & \vdots & \ldots & 1 \end{array} \right) $$
$Σ_r(ρ_r)$回归矩阵具体如下:
$$ \left( \begin{array}{ccc} 1 & ρ_r & ρ_r^2 & \ldots & ρ_r^{r-1} \\ ρ_r & 1 & ρ_r & \ldots & \vdots \\ ρ_r^2 & ρ_r & 1 & \ldots & \vdots \\ \vdots & \vdots & \vdots & \ddots & \vdots \\ ρ_r^{r-1} & \vdots & \vdots & \ldots & 1 \end{array} \right) $$
假定η随机误差是独立的,则误差方差协方差矩阵为: $$Var(e) = \sigma_\xi^2 [Σ_c(ρ_c) \otimes Σ_r(ρ_r)] + \sigma_\eta^2I$$
其中,$\sigma_\xi^2$是空间相关的误差,$\sigma_\eta^2$是独立的误差,I是单位矩阵。
数学公式不少,部分读者估计已经懵了。空间分析原理通俗点说,正如古人所言的‘近朱者赤,近墨者黑’,拿试验林为例,就是部分林地内树木都表现良好,而部分林地下却总体表现差,于是问题就来了,这些局部表现优秀或低劣的树木,到底是因为所处的微环境(比如光照、土壤养分等)还是其自身的基因型造成的?这里面事实上有两种情况:一种是基因型优秀的,但运气欠佳,落到了贫瘠的土壤里,成了‘怀才不遇型’;另一种是基因型低劣的,但运气爆棚,落到了肥沃的土壤里,成了‘滥竽充数型’。因此,空间分析就是要纠正这两种情况,通过空间自相关或者其它途径。有趣的是,基因型优秀或者低劣,是相对于试验环境而言的,即某个基因型在一种试验环境中是优秀的,但到了另一种试验环境中可能就成了低劣型。这属于基因型与环境互作的问题,本文不作这方面的分析。
现在来看看在R中如何使用breedR包进行空间分析的演示,虽然asreml包更强大,但由于它是商业软件,且费用不低,因此本文不打算介绍asreml。
2 空间模型的分析示例
首先载入所需程序包
|
|
(2.0) 示例数据集
示例采用agridat包的数据集stroup.nin,该数据含有小麦基因型gen、区组rep、产量yield、列号col和行号row等5个变量。
|
|
数据集的变量及其特征:
|
|
目标性状yield的直方图(Fig.1)和空间趋势(Fig.2)如下:
|
|
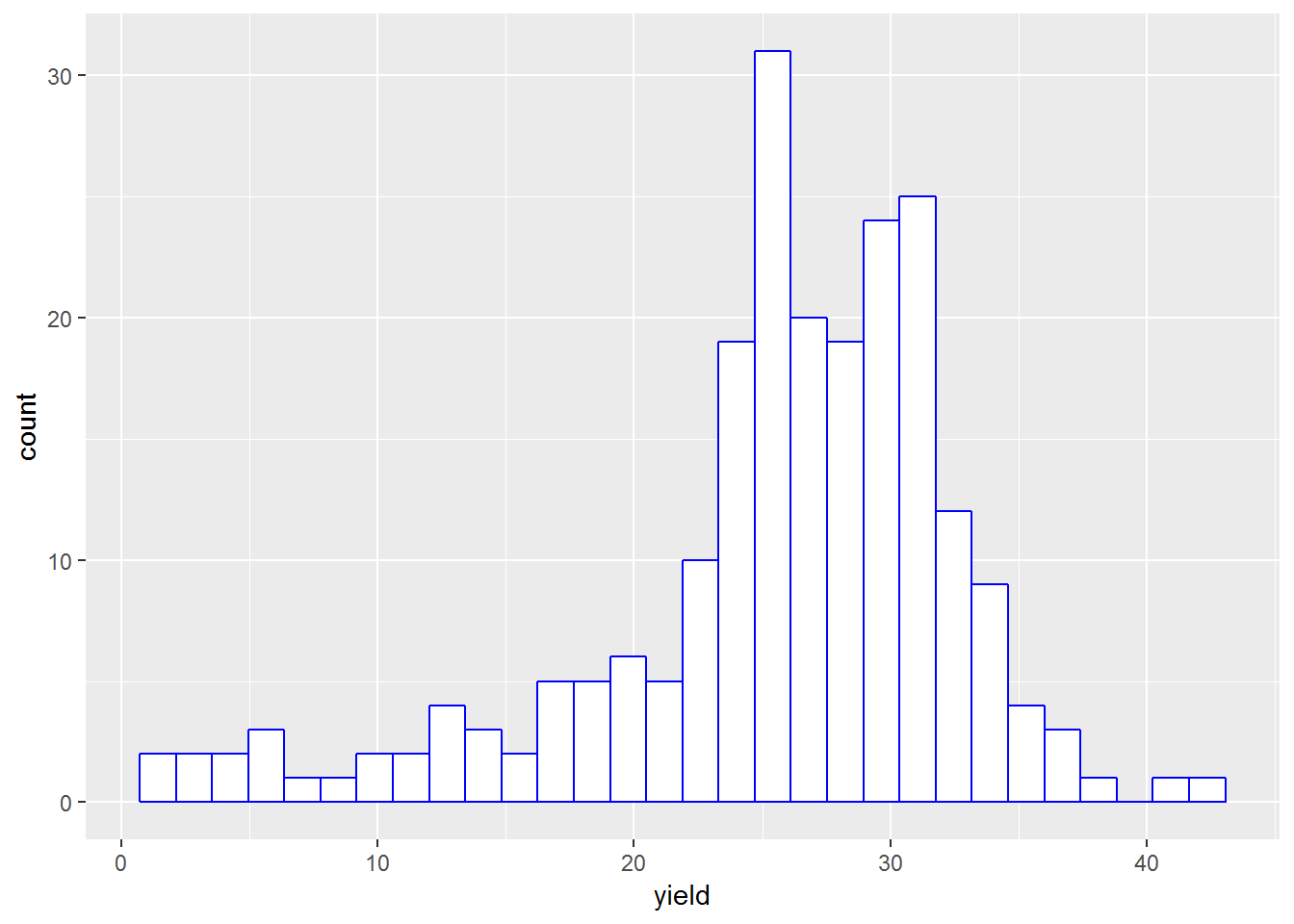
目标性状yield的直方图
|
|
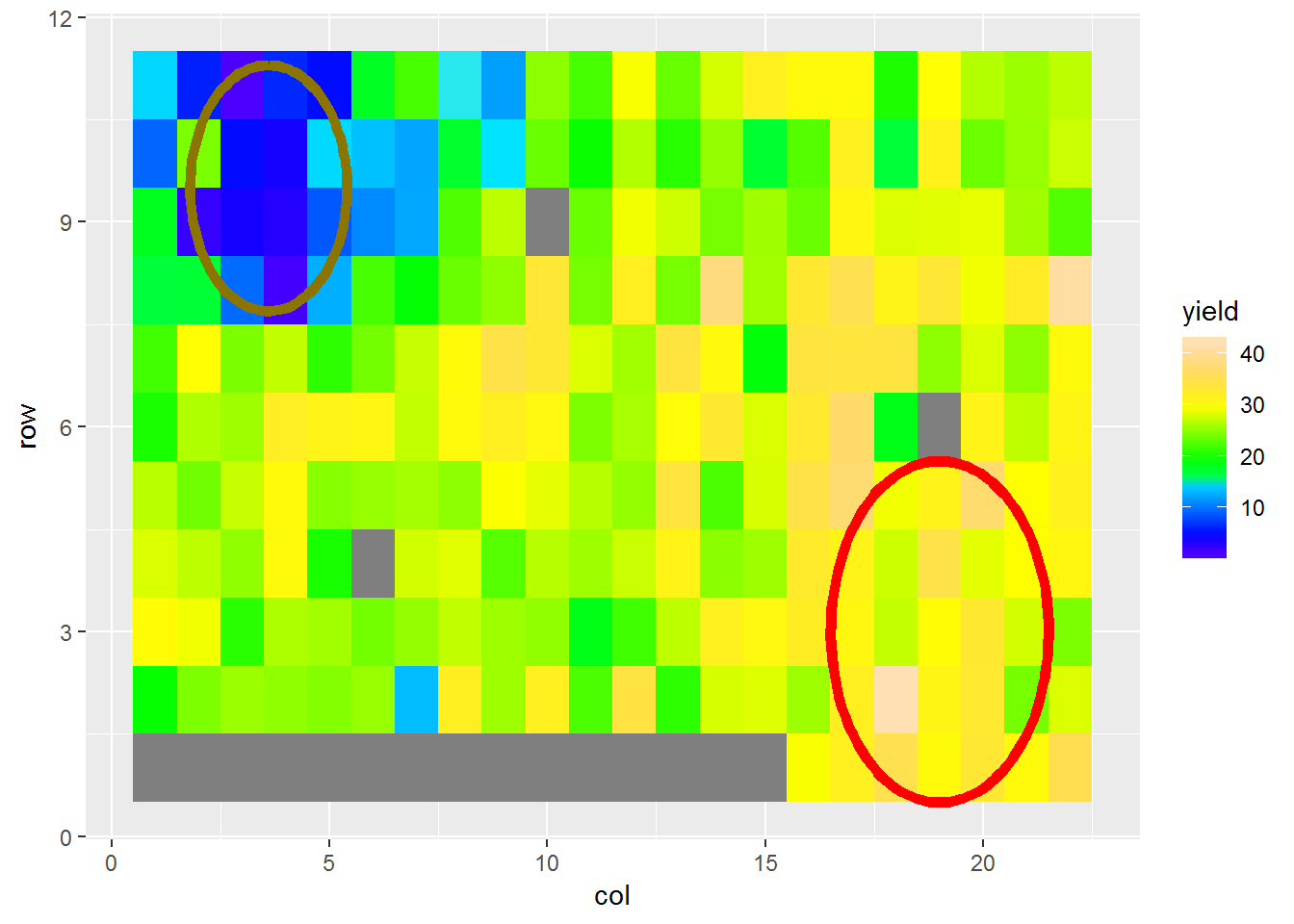
目标性状yield的空间趋势
(2.1)分析模型
(2.1.1) 模型1–RCB模型
首先采用原本的试验设计–随机完全区组设计(RCB),进行数据分析。由于数据要针对基因型的品种表现作评估与比较,因此将其作为固定效应。具体代码如下:
|
|
RCB模型的方差分量以及Loglook与AIC值如下:
|
|
此外,breedR允许将区组放入空间分析方法中,但结果与RCB的一样。分析代码如下:
|
|
RCB1模型的方差分量以及Loglik与AIC值如下:
|
|
(2.1.2) 模型2–SUM空间模型
现在进行空间模型分析,由于breedR包不能设定,$\sigma_\eta^2$=0,因此它无法进行仅含有空间相关误差的模型,这与asreml相比,多少有点遗憾,但对于初学者,这却是优点!下面展示行列均相关的空间分析模型,具体代码如下:
|
|
SUM模型的方差分量以及Loglik与AIC值如下:
|
|
SUM模型的空间相关误差趋势图如下:
|
|
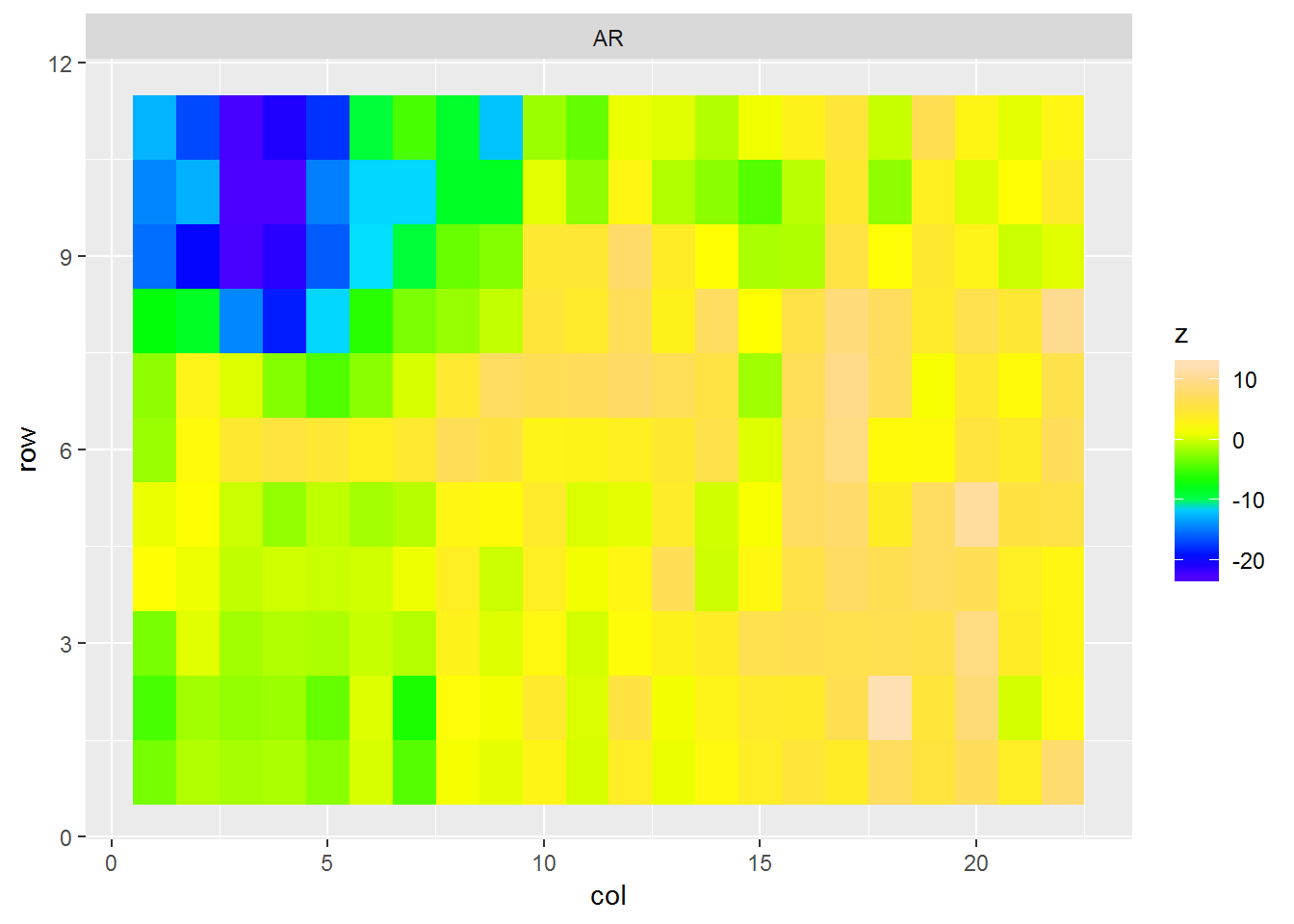
SUM模型的空间相关误差趋势图
(2.1.3) 模型3–ColAR空间模型
现在设定只有列存在空间相关,即行之间不存在自相关。代码如下:
|
|
ColAR模型的方差分量以及Loglik与AIC值如下:
|
|
ColAR模型的空间相关误差趋势图如下:
|
|
(2.1.4) 模型4–RowAR空间模型
现在设定只有行存在空间相关,即列之间不存在自相关。代码如下:
|
|
RowAR模型的方差分量以及Loglik与AIC值如下:
|
|
RowAR模型的空间相关误差趋势图如下:
|
|
(2.2) 四种模型的空间相关误差比较
注意:RCB1模型是把区组放到空间分析方法里,因此所计算的空间相关误差其实就是区组方差。
|
|
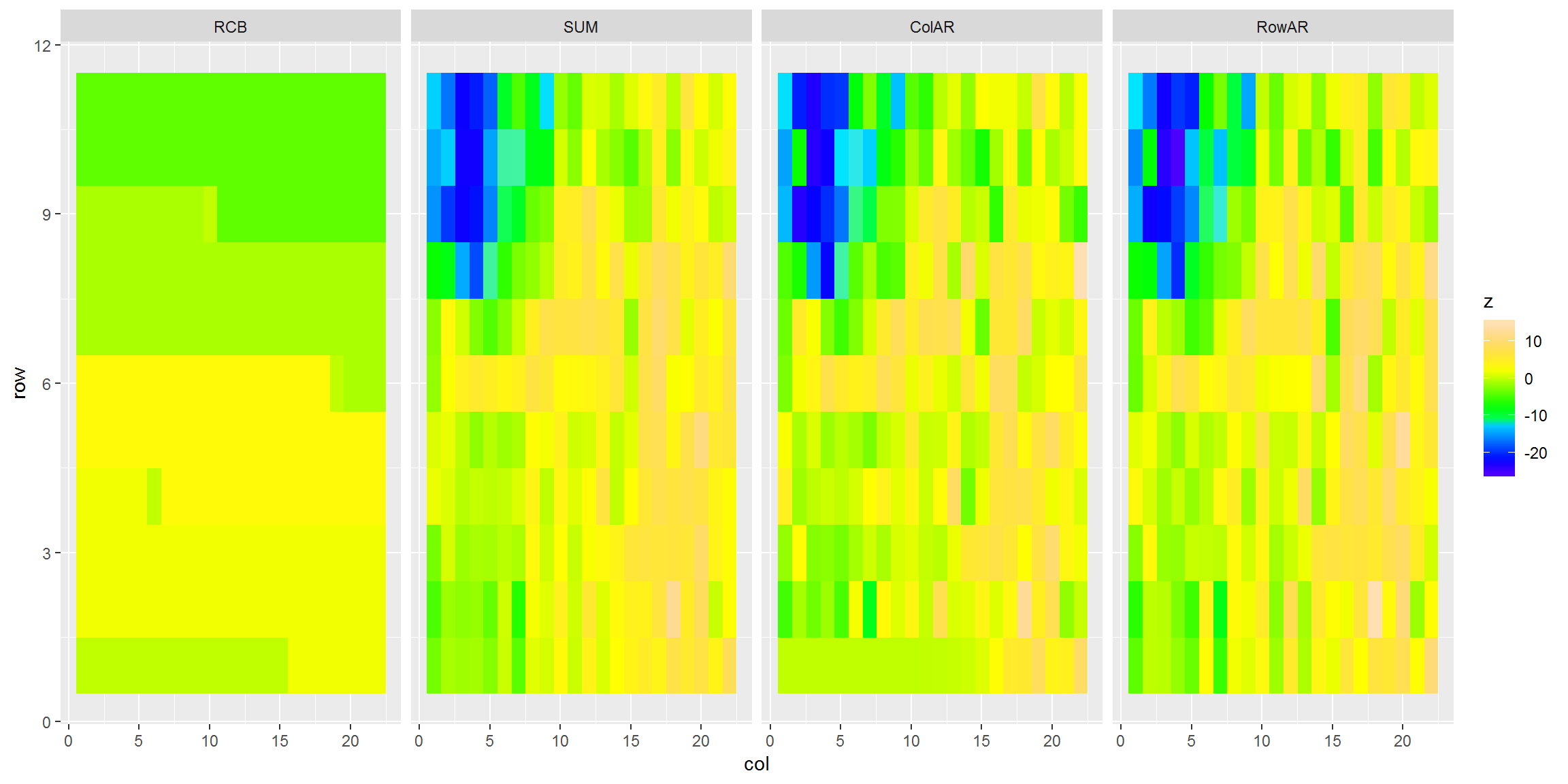
四种模型的空间相关误差趋势图
从图4可以明显看出空间模型与RCB模型的图形较大,虽然3种空间模型的变化比较小,但认真查看,还是能看到微小的差异。此外,对于空间分析,还有variogram(半残差)图,但因为breedR包提供的variogram图实在太丑了,这里不做演示。
(2.3) 四种模型的拟合统计量比较
|
|
| Model | AIC | logLik | Spatial | Residual |
|---|---|---|---|---|
| RCB | 1253.938 | -624.969 | 9.883 | 49.582 |
| SUM | 1147.881 | -571.941 | 35.757 | 5.536 |
| ColAR | 1198.539 | -597.269 | 43.711 | 0.002 |
| RowAR | 1198.989 | -597.495 | 57.039 | 0.003 |
从统计量结果可知,对于含有空间相关的模型,不论是行列双向自相关还是行或列单向自相关,模型拟合的效果都要优于RCB模型,因为它们的AIC值都小于RCB模型。其实,这不难理解,因为传统的RCB模型,区组往往比较大,而在空间分析模型里,通过行列自相关模型,其实是把区组细化到小区(每个行列组合),因此模型所估计的误差方差更为精确(例如RCB模型和SUM模型,随机误差方差已从49.582下降到5.536!),当然AIC值就会变小。根据AIC准则,SUM模型是本例中的最优模型,即行列双向自相关。
(2.4) 四种模型的gen效应值比较
|
|
通过上述代码获取各模型的固定gen效应值,然后按最优模型M2的效应值降序排序,并输出结果的前8名。
|
|
| ID | m1 | m1.se | m1.rank | m2 | m2.se | m2.rank | m3 | m3.se | m3.rank | m4 | m4.se | m4.rank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NE86503 | 32.65 | 3.856 | 1 | 26.49 | 2.473 | 14 | 29.33 | 2.846 | 5 | 27.56 | 2.632 | 11 |
| NE87619 | 31.26 | 3.856 | 2 | 29.43 | 2.552 | 2 | 29.84 | 2.849 | 2 | 30.00 | 2.720 | 2 |
| NE86501 | 30.94 | 3.856 | 3 | 25.13 | 2.554 | 27 | 28.42 | 2.904 | 11 | 25.56 | 2.752 | 22 |
| Redland | 30.50 | 3.856 | 4 | 28.41 | 2.574 | 5 | 29.54 | 2.891 | 3 | 28.45 | 2.820 | 6 |
| Centurk78 | 30.30 | 3.856 | 5 | 26.39 | 2.584 | 16 | 28.07 | 2.897 | 13 | 26.60 | 2.724 | 12 |
| NE83498 | 30.12 | 3.856 | 6 | 29.34 | 2.609 | 3 | 29.41 | 2.882 | 4 | 29.85 | 2.850 | 3 |
| Siouxland | 30.11 | 3.856 | 7 | 23.89 | 2.526 | 37 | 28.01 | 2.895 | 14 | 24.11 | 2.647 | 36 |
| NE86606 | 29.76 | 3.856 | 8 | 27.43 | 2.503 | 9 | 28.42 | 2.850 | 10 | 28.12 | 2.658 | 7 |
|
|
| ID | m1 | m1.se | m1.rank | m2 | m2.se | m2.rank | m3 | m3.se | m3.rank | m4 | m4.se | m4.rank |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buckskin | 25.56 | 3.856 | 28 | 33.78 | 2.531 | 1 | 31.61 | 2.941 | 1 | 32.11 | 2.710 | 1 |
| NE87619 | 31.26 | 3.856 | 2 | 29.43 | 2.552 | 2 | 29.84 | 2.849 | 2 | 30.00 | 2.720 | 2 |
| NE83498 | 30.12 | 3.856 | 6 | 29.34 | 2.609 | 3 | 29.41 | 2.882 | 4 | 29.85 | 2.850 | 3 |
| NE85556 | 26.39 | 3.856 | 23 | 28.98 | 2.559 | 4 | 28.82 | 2.844 | 8 | 28.76 | 2.732 | 5 |
| Redland | 30.50 | 3.856 | 4 | 28.41 | 2.574 | 5 | 29.54 | 2.891 | 3 | 28.45 | 2.820 | 6 |
| Brule | 26.07 | 3.856 | 25 | 28.08 | 2.553 | 6 | 26.28 | 2.888 | 22 | 29.20 | 2.734 | 4 |
| NE86507 | 23.79 | 3.856 | 39 | 27.67 | 2.473 | 7 | 26.36 | 2.844 | 20 | 27.77 | 2.654 | 9 |
| Scout66 | 27.52 | 3.856 | 17 | 27.53 | 2.554 | 8 | 28.82 | 2.843 | 7 | 25.91 | 2.777 | 17 |
从输出结果看,OMG,结果相差甚远!尤其是RCB模型与最优模型SUM之间,基本完全不同,例如SUM中最好的基因型Buckskin在模型RCB仅排名第28名,而且产量被RCB模型严重低估了!本例中,基因型Buckskin在RCB模型中就是‘怀才不遇型’;基因型NE86503在RCB模型中就是‘滥竽充数型’,合理的空间分析模型给予了纠正。即便是3种空间模型,它们之间的差异也较大。此外,最优模型的标准误se也最小!这结果验证了空间分析的优越性,也验证了统计分析方法的重要性!
空间分析结果对人生的提示:只要是金子,迟早会发光,即便一段时间内怀才不遇;如果是狐假虎威,迟早会败露,即便一段时间内风光无限。
3 结语
3.1 从本例的分析结果来看,选择最好的软件非常有必要!此外,关注新的统计方法也非常关键!本例结果显示SUM是最优模型,那么它有没再改进的可能?答案是肯定的,感兴趣的读者可以阅读本人的一篇文章3。
3.2
空间分析的使用条件。诚如已有菜谱,但能否每人都根据菜谱做出可口的上等好菜,这需要磨练和时间。同理,空间分析模型虽好,也得要知道它的使用条件和判断标准。首先,数据集里必须有行列号;其次,对于简单数据集,模型不可过于复杂,否则可能会造成过拟合(overfit);再者,合理的空间模型可能并唯一,但不合理的空间模型可能会导致结果高估或低估。
后话:虽然空间分析已成为农林业国际的常用方法之一,但其在国内林业遗传评估上的应用仍然有限!我最近搜了搜关于数量遗传学的著作或教材,国内外也是更新极慢,经典当属《Intrudction to quantitative genetics(4th)》(1996年)和《Genetics and Analysis of Quantitative Traits》(1998年),历经20年了!第一本经典的作者Douglaus Falconer,可惜已于2004年逝世。第二本的作者今年出版第二部《Evolution and Selection of Quantitative Traits》,9月份出版!其实,关于统计分析史的那些大师们,有些故事还是非常耐人寻味的!多少有点遗憾,迷人的统计故事都是国外的大师造就的。
参考资料
- https://www.zhihu.com/question/28409028 [return]
- 林元震主编.《R与ASReml-R统计学》.中国林业出版社.2017 [return]
- http://blog.sciencenet.cn/blog-1114360-1071888.html [return]
