巧用plyr包可以实现很多情况下的批量分析。如果手头经常需要重复运行某些类似程序,就可以编个函数和dplyr包一起使用,达到事半功倍。
(1)使用plyr包思路的来源
在我主编的《R与ASReml-R统计学》第422~424页,演示了在ggplot2包绘制的分面图上批量添加回归方程,具体例子如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
|
lm_labels <- function(dat) {
mod <-lm(h5 ~ h3, data=dat)
formula <-sprintf("italic(y) == %.2f %+.2f * italic(x)",
round(coef(mod)[1],2), round(coef(mod)[2],2))
r <-cor(dat$h5,dat$h3,use="pairwise")
r2 <- sprintf("italic(R^2) == %.2f", r^2)
data.frame(formula=formula,r2=r2,stringsAsFactors=FALSE)
}
# library(plyr)
data(dfm,package='RSTAT2D')
labels <- plyr::ddply(dfm,"Spacing",lm_labels)
labels <- lm_labels(dfm)
# library(ggplot2)
ggplot(dfm,aes(x=h3,y=h5))+ geom_point() +
facet_wrap(~Spacing, labeller=label_both)+
geom_smooth(method=lm, se=TRUE,fill='red',alpha=.3) +
geom_text(x=250,y=450,aes(label=formula), data=labels, parse=TRUE,hjust=0) +
geom_text(x=250,y=415,aes(label=r2),data=labels,parse=TRUE,hjust=0)
|
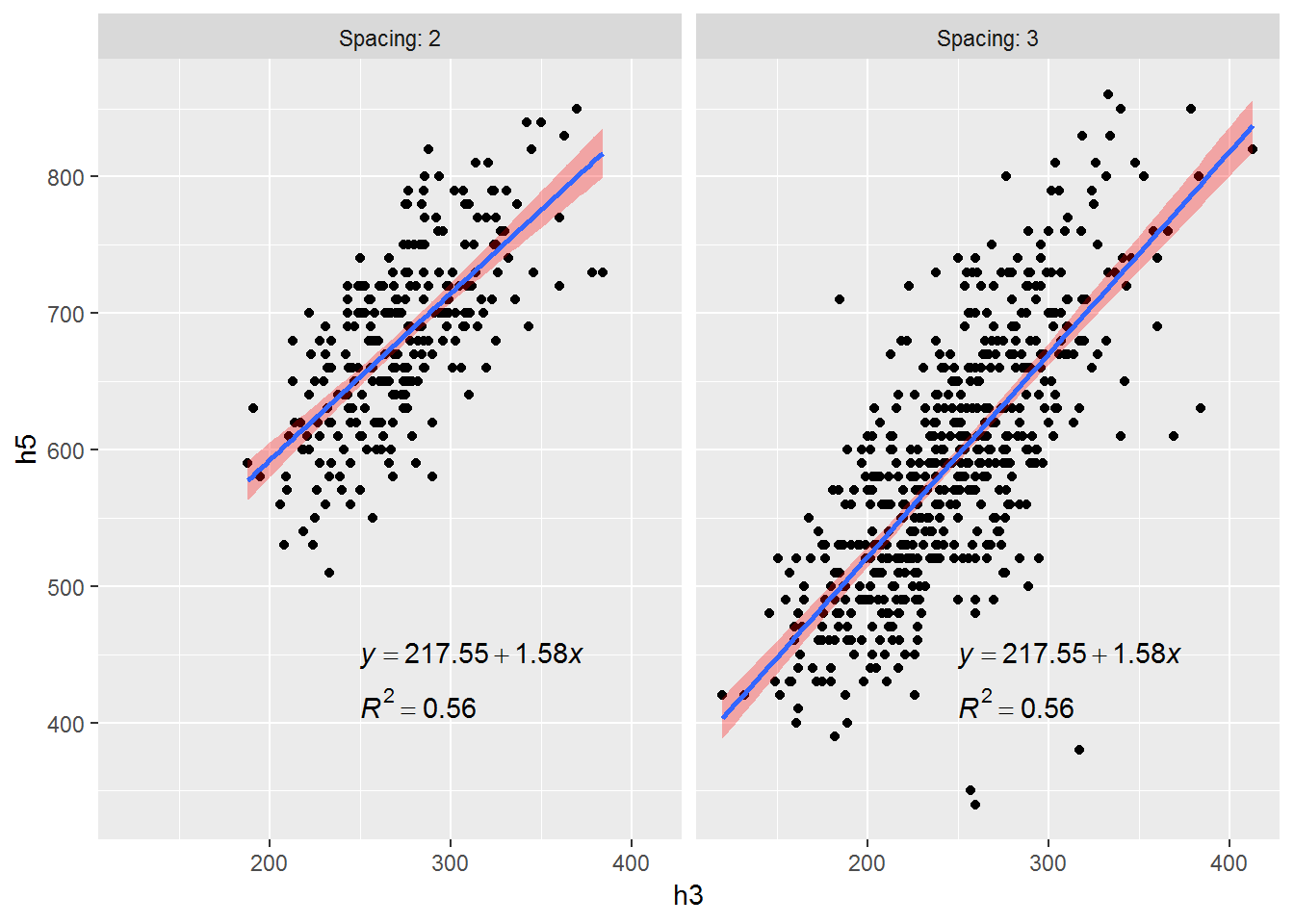
上述图形实现的思路是,编一个函数lm_labels()进行目标数据的线性回归并返回线性方程及其R2,然后通过plyr包的ddply()对分组变量或因子Spacing进行函数lm_labels()的运算,最后返回一个数据框labels。labels含有注释所需的所有信息,再通过geom_text()添加到图形中。
那么能否运用到模型的批量计算呢?从思路上来说,应该行得通。于是就试试混合线性模型。结果是,当然可以运行。
(2) plyr包进行线性混合模型的批量分析
下文将使用nlme、lme4、asreml和breedR包分别进行演示,部分代码在上述程序包均为相似,因此先编一个小函数,用于数据的变换,即2行
× N行的数据框转换为1行排列。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
# function change data frame (2 X N) into one line
mf.tr <- function(df) {
df<-as.data.frame(df)
df<-df[sort(names(df))]
nn<-ncol(df)
res.ls<-data.frame(ls=NA)
res.ls[,1:nn]<-round(df[1,],3)
res.ls[,(nn+1):(2*nn)]<-round(df[2,],3)
NVar<-colnames(df)
NVar.se<-paste(NVar,'se',sep='.')
names(res.ls)<-c(NVar,NVar.se)
return(res.ls)
}
|
(2.0) 示例数据集
示例采用agridat包的数据集butron.maize,该数据含有个体、双亲、地点和产量等5个变量,为了批量分析,特意模拟一个正态分布的变量x。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
|
data(butron.maize,package='agridat')
str(butron.maize)
## 'data.frame': 245 obs. of 5 variables:
## $ gen : Factor w/ 49 levels "A509xA637",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ male : Factor w/ 9 levels "A509","A637",..: 1 1 1 1 1 1 1 1 1 2 ...
## $ female: Factor w/ 9 levels "A637","A661",..: 1 2 3 4 5 6 7 8 9 2 ...
## $ env : Factor w/ 5 levels "pc95","pc96",..: 3 3 3 3 3 3 3 3 3 3 ...
## $ yield : num 6.81 5.02 7.39 6.84 6.04 7.1 4.37 5.89 4.48 7.02 ...
butron.maize1<-tidyr::unite(butron.maize,col=Fam,2:3,sep='.')
butron.maize2<-dplyr::left_join(butron.maize,butron.maize1,
by=c('gen','env','yield'))
set.seed(2018)
butron.maize2$x<-rnorm(245,mean=10,sd=4)
# 将需要的分析性状yield和x整合到变量Trait下,
# 将yield和x对应的数值整合到变量y下,
# 以便使用plyr的ddply()通过Trait进行批量分析
df<-tidyr::gather(butron.maize2,key=Trait,y,c(-1:-4,-6))
str(df)
## 'data.frame': 490 obs. of 7 variables:
## $ gen : Factor w/ 49 levels "A509xA637",,..: 1 2 3 4 5 6 7 ...
## $ male : Factor w/ 9 levels "A509","A637",..: 1 1 1 1 1 1 1 1 1 2 ...
## $ female: Factor w/ 9 levels "A637","A661",..: 1 2 3 4 5 6 7 8 9 2 ...
## $ env : Factor w/ 5 levels "pc95","pc96",..: 3 3 3 3 3 3 3 3 3 3 ...
## $ Fam : chr "A509.A637" "A509.A661" "A509.CM105" "A509.EP28" ...
## $ Trait : chr "yield" "yield" "yield" "yield" ...
## $ y : num 6.81 5.02 7.39 6.84 6.04 7.1 4.37 5.89 4.48 7.02 ...
|
(2.1) 与nlme包结合使用
首先,编写nlme包做线性混合分析的通用函数,具体代码如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
|
nlme.batch <- function(data,FMod,RMod){
# nlme V3.1-127
nlm<-nlme::lme(FMod,random=RMod,data=na.omit(data))
Var<-VarCorr(nlm)
suppressWarnings(storage.mode(Var) <- "numeric")
vc<-Var[!is.na(Var)]
Var1<-matrix(vc,nrow=2,byrow=T)
RFN<-rownames(Var)
RFN<-RFN[RFN!="(Intercept)"]
RFN1<-gsub(" =",'',RFN)
colnames(Var1)<-RFN1
res.ls<-mf.tr(Var1)
return(res.ls)
}
|
现在使用已经构建的数据集df和函数nlme.batch()来试试批量分析,当然使用函数nlme.batch()前,需要指定模型的固定效应和随机效应,具体示例如下:
1
2
3
4
5
6
7
8
9
|
# 固定效应
Fixed.Mod= y ~ 1+env
# 随机效应
Ran.Mod=~1|male/female/Fam
#library(nlme)
sp1<-plyr::ddply(df,'Trait',
function(df) nlme.batch(data=df,FMod=Fixed.Mod,RMod=Ran.Mod))
|
固定项Fixed.Mod= y ~ 1+env,因为目标变量yield和x的数值现在都已整合入变量y中。随机项Ran.Mod=~1|male/female/Fam表示含有3个随机效应,分别为male、female和Fam。由于nlme无法直接识别male:female,因此利用tidyr包的unite()创建变量Fam,指定其为male和female的组合。这样处理数据时,应当注意双亲是完全不同的。本例中,这样处理没有问题,原因如下:
1
2
3
4
5
6
7
8
9
10
11
12
|
table(df$male,df$female)
##
## A637 A661 CM105 EP28 EP31 EP42 F7 PB60 Z77016
## A509 10 10 10 10 10 10 10 10 10
## A637 0 10 10 10 10 10 10 10 10
## A661 0 0 10 10 10 10 10 10 10
## CM105 0 0 0 10 10 10 10 10 10
## EP28 0 0 0 0 10 10 10 10 10
## EP31 0 0 0 0 0 10 10 10 10
## EP42 0 0 0 0 0 0 10 10 10
## F7 0 0 0 0 0 0 0 10 10
## PB60 0 0 0 0 0 0 0 0 10
|
因为nlme.batch()函数返回数据框,无法直接查看结果,因此再运行sp1,即可看到方差分量结果:
1
2
3
4
|
sp1
## Trait Fam female male Residual Fam.se female.se male.se Residual.se
## 1 x 0.270 0.270 0.359 15.466 0.520 0.520 0.599 3.933
## 2 yield 0.231 0.231 0.493 0.570 0.481 0.481 0.702 0.755
|
(2.2) 与lme4包结合使用
首先,编写lme4包做线性混合分析的通用函数,具体代码如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
|
lme4.batch <- function(data,Mod){
# lme4 V1.1-13
lme<-lme4::lmer(Mod,data=data)
Var<-as.data.frame(summary(lme)$varcor)[,-2:-3]
row.names(Var)<-Var[,1]
Var<-Var[,-1]
Var<-as.data.frame(t(Var))
res.ls<-mf.tr(Var)
return(res.ls)
}
|
现在使用函数lme4.batch()来试试批量分析,但需要注意lme4指定模型的固定效应和随机效应不同于多数R包的格式,其并未将固定效应与随机效应分开,且以(1|male)表示随机效应,具体示例如下:
1
2
3
4
5
6
7
|
# 固定效应和随机效应
Mod= y ~ 1+env+(1|male)+(1|female)+(1|Fam)
#Mod= y ~ 1+env+(1|male)+(1|female)+(1|male:female)
# library(lme4)
sp2<-ddply(df,'Trait',
function(df) lme4.batch(data=df,Mod))
|
因为lme4.batch()函数返回数据框,无法直接查看结果,因此再运行sp2,即可看到方差分量结果:
1
2
3
4
|
sp2
## Trait Fam female male Residual Fam.se female.se male.se Residual.se
## 1 x 0.540 0.000 0.359 15.466 0.735 0.000 0.599 3.933
## 2 yield 0.194 0.256 0.587 0.570 0.440 0.506 0.766 0.755
|
(2.3) 与asreml包结合使用
首先,编写asreml包做线性混合分析的通用函数,具体代码如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
|
asr0.batch <- function(data,FMod=NULL,RMod=NULL,EMod=NULL){
if(is.null(EMod)) EMod=~ units
# asreml V3.0
asr<-asreml::asreml(fixed=FMod, random=RMod, rcov=EMod,
na.method.X='include',
data=data, trace=FALSE)
Var<-t(summary(asr)$varcomp[,2:3])
Var<-as.data.frame(Var)
res.ls<-mf.tr(Var)
return(res.ls)
}
|
现在使用函数asreml()来试试批量分析,具体示例如下:
1
2
3
4
5
6
7
8
|
FMod1=y~ 1+env
RMod1=~male+female+Fam
#RMod1=~male+female+male:female
library(asreml)
sp3<-ddply(df,'Trait',
function(df) asr0.batch(data=df,FMod=FMod1,
RMod=RMod1))
|
因为asr0.batch()函数返回数据框,再运行sp3,即可看到方差分量结果:
1
2
3
4
5
6
7
|
sp3
## Trait Fam!Fam.var female!female.var male!male.var R!variance
## 1 x 0.386 0.000 0.357 15.771
## 2 yield 0.204 0.267 0.578 0.581
## Fam!Fam.var.se female!female.var.se male!male.var.se R!variance.se
## 1 0.823 0.000 0.576 1.593
## 2 0.085 0.179 0.348 0.059
|
由于asreml对结果的注释比较仔细,因此结果看起来不太好看,可以再进行对上述结果做命名的变换,示范如下:
1
2
3
4
5
6
7
8
9
|
nn<-names(sp3)
nn1=nn2=NULL
for(i in 1:5) {
nn1[i]<-strsplit(nn[i],'!')[[1]][1]
nn2[i]<-nn1[[i]][1]
}
nn2<-nn2[-1]
nn2.se<-paste(nn2,'se',sep='.')
names(sp3)[-1]<-c(nn2,nn2.se)
|
现在结果格式与之前的两个包相似。关于asreml返回变量名称的修改,更明智的做法是在asr0.batch()函数进行修改,因为它是通用函数。
1
2
3
4
5
6
7
|
sp3
## Trait Fam!Fam.var female!female.var male!male.var R!variance
## 1 x 0.386 0.000 0.357 15.771
## 2 yield 0.204 0.267 0.578 0.581
## Fam!Fam.var.se female!female.var.se male!male.var.se R!variance.se
## 1 0.823 0.000 0.576 1.593
## 2 0.085 0.179 0.348 0.059
|
下述是我修改后的通用函数asr1.batch(),就可得到比较直观的结果:
1
2
3
4
5
6
7
8
9
10
|
sp3b<-ddply(df,'Trait',
function(df) asr1.batch(data=df,FMod=FMod1,RMod=RMod1))
sp3b
## Trait loglik AIC male female Fam Rvar male.se female.se
## 1 x -464.990 937.980 0.357 0.000 0.386 15.771 0.576 0.000
## 2 yield -102.554 213.108 0.578 0.267 0.204 0.581 0.348 0.179
## Fam.se Rvar.se converge
## 1 0.823 1.593 TRUE
## 2 0.085 0.059 TRUE
|
综上所述,巧用plyr包可以实现很多情况下的批量分析。如果手头经常需要重复运行某些类似程序,就可以编个函数和dplyr包一起使用,达到事半功倍。虽然,上述程序看起来还算不错,但遗憾的是,好像不能用于多性状的批量分析,即只能用于单性状的批量分析。asreml的多性状批量分析,还是只能使用我的老办法asreml.batch,对此感兴趣者可以查看本人科学网博客。

